共生微生物通过诱导角质形成细胞HIF-1α信号通路和谷氨酰胺代谢促进毛囊再生
点评理由:该研究创新性强,结果客观全面,结论具有临床指导意义。对于皮肤毛囊再生的治疗具有重要意义。谷氨酸或IL-1β的添加,可能促进患者伤口愈合,而局部抗生素的使用,则有可能削弱常驻菌群的再生作用。
微生物在组织内稳态维持、愈合和再生中起着重要作用,皮肤共生微生物对宿主的代谢调节必不可少[1]。既往研究证明,人体皮肤的共生微生物,对皮肤损伤再生有积极作用,特别是通过白细胞介素-1β(IL-1β)信号传导[2];然而,再生过程中,微生物相关的具体作用机制尚不清楚。
共生微生物发挥怎样的作用?再生过程发生了何种代谢改变?谷氨酰胺在其中发挥了怎样的作用?缺氧诱导因子-1α(HIF-1α)信号通路又发挥了怎样的作用?学者们希望将这些未连接因素,统一到一个连贯的生理模型中,来解释微生物促进皮肤毛囊再生的过程。
近日,南方医科大学南方医院王高峰团队以及约翰霍普金斯大学医学院Luis A. Garza团队在《SCIENCE ADVANCES》期刊发表了重要的研究成果。他们在创伤性毛囊再生(WIHN,wound-induced hair follicle neogenesis)的器官再生模型中发现,皮肤共生微生物组诱导创面微环境缺氧,激活HIF-1α信号通路,促进角质形成细胞内谷氨酰胺代谢,进而激活下游IL-1β信号通路,促进WIHN。这一发现对于,促进皮肤和毛囊再生具有重要的治疗意义。
WIHN指的是皮肤创伤性毛囊再生过程。研究人员选择了金黄色葡萄球菌作为共生微生物的代表,首先探究了细菌负荷与WIHN之间的关系,结果显示,细菌负荷与WIHN呈剂量反应性相关。分类分析结果显示,皮肤微生物通过角化形成细胞依赖的IL-1β-Myd88信号诱导了WIHN。这说明了共生微生物、IL-1β、WIHN过程之间的密切联系。
接下来,研究人员想要探究细菌如何诱导IL-1β促进再生。代谢组学结果显示,在高细菌负荷和高WIHN的创面中,谷氨酰胺代谢是显著增加的(图1)。
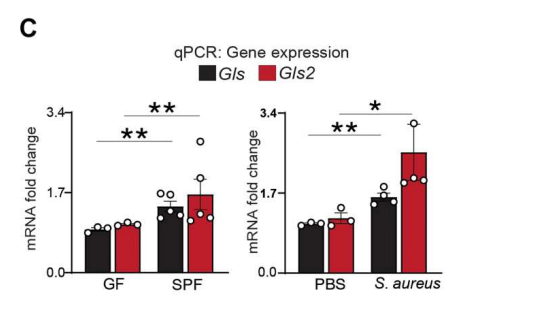
图1. 细菌诱导伤口创面的谷氨酰胺代谢
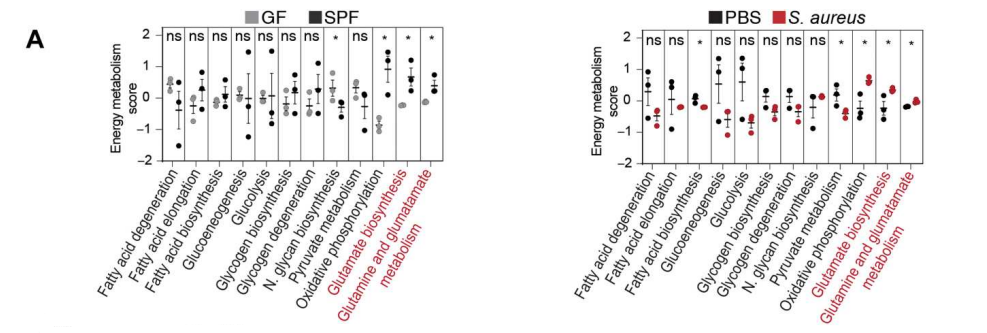
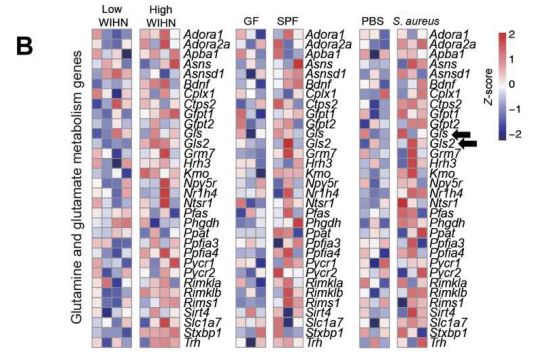
(A)能量代谢评分;(B)谷氨酰胺和谷氨酸基因标记微阵列图
(C)定量逆转录聚合酶链反应(qRT-PCR)检测到的小鼠中谷氨酰胺酶的mRNA表达
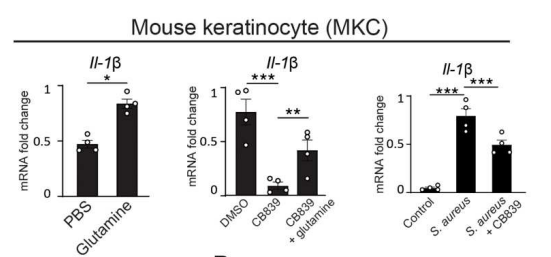
为了确定谷氨酰胺代谢是否影响角质形成细胞中IL-1β的表达,从而促进再生,研究人员分离、培养并用谷氨酰胺处理小鼠角质形成细胞。体外研究结果显示,谷氨酰胺促进角质形成细胞中IL-1βmRNA的表达。谷氨酰胺酶抑制剂CB839处理的角质形成细胞IL-1β表达则下降,通过加入外源性谷氨酰胺可以挽救(图2)。
图2. 细菌通过谷氨酰胺代谢产生IL-1β来诱导角化生成细胞
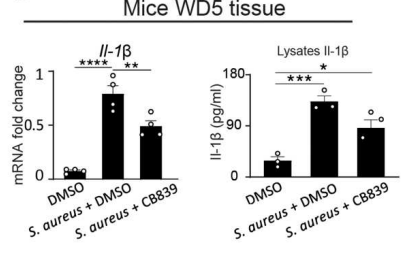
随后,作者进一步研究了谷氨酰胺代谢对IL-1β产生的体内影响。结果显示,金黄色葡萄球菌能够有效诱导IL-1β蛋白和mRNA产生,CB839则会产生抑制效应(图3)。在人角质形成细胞中的结果类似。这些结果表明,皮肤细菌,特别是金黄色葡萄球菌,促进了角质形成细胞谷氨酰胺代谢,谷氨酰胺代谢对角质形成细胞IL-1β的产生至关重要。
图3. 小鼠体内金黄色葡萄球菌有效诱导IL-1β蛋白和mRNA产生
研究者还进行了纳入健康成年志愿者的人体试验。研究方法是,在人体双侧皮肤进行穿刺活检伤口,然后分别使用凡士林或局部抗生素(Neosporin)进行治疗。结果显示皮肤的创伤会诱导代谢模式的较大转变,谷氨酰胺代谢在创伤皮肤中被高度诱导。抗生素则显著降低了伤口愈合速度,并改变了微生物组。这些数据表明,人类皮肤上的共生细菌可以诱导谷氨酰胺代谢,改善再生,这与我们的体外结果一致。
接下来,研究人员进行了谷氨酰胺代谢诱导体外干细胞标志物表达和再生信号的研究。体外研究结果显示,谷氨酰胺通过IL-1β-MyD88信号诱导再生相关基因,说明IL-1β是谷氨酰胺诱导再生基因表达的下游信号。
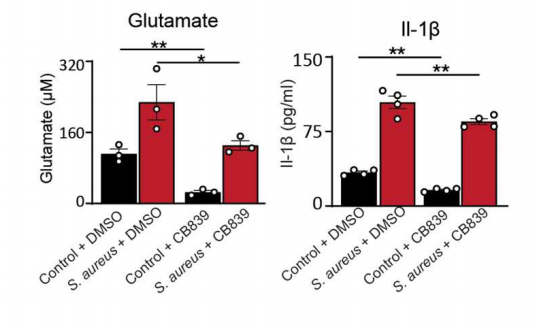
为了更全面地了解再生过程。作者进一步研究了谷氨酰胺代谢对体内毛囊再生的影响。结果显示,谷氨酸盐在再生的基线阶段和细菌诱导的后续阶段都十分重要。另外,细菌诱导的毛囊再生依赖于谷氨酰胺代谢,并且谷氨酰胺代谢通过角质形成细胞中IL-1β依赖性Myd88信号促进毛囊再生。(图4)
图4. 谷氨酸是基线阶段和细菌诱导的WIHN所必需的
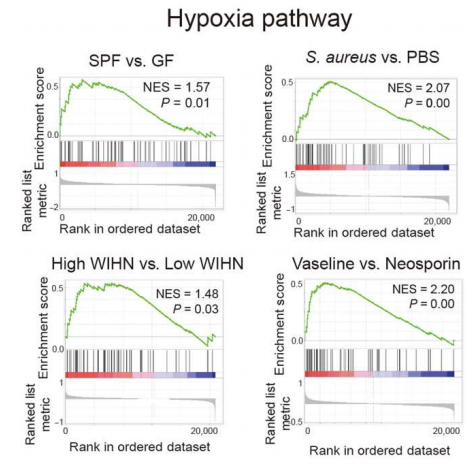
既往研究结果显示,细菌负荷增加可诱导皮肤缺氧,而缺氧诱导的HIF-1α信号促进谷氨酰胺代谢和IL-1β合成[3]。研究人员进行了缺氧环境与细菌促进皮肤再生、谷氨酰胺、IL-1β等之间的机制关系。结果显示,大多数缺氧相关基因在高再生的伤口中高表达,高再生与低氧转录组特征具有相关性(图5);体外低氧条件下角质形成细胞谷氨酰胺代谢和IL-1β的变化,与正常氧条件下一致,并且CB839在2%氧气下可抑制IL-1β的表达。另外,敲低HIF-1α,可以抑制低氧条件下培养的角质形成细胞的IL-1β表达。以上结果说明,细菌通过缺氧诱导的HIF-1α信号促进谷氨酰胺代谢,进而诱导IL-1β产生和再生信号,从而促进WIHN。
图5. 细菌通过缺氧诱导的HIF信号刺激谷氨酰胺代谢和IL-1β的产生-高再生与低氧转录组特征具有相关性
为进一步对结论进行验证。作者团队区分正常皮肤、小伤口、大伤口中心(WIHN)、大伤口周边(非WIHN)对WIHN过程中缺氧和谷氨酰胺代谢进行了研究。结果显示,相比于外周,大伤口中心(WIHN)的角质形成细胞,特别是再生毛囊,需要更多的能量和谷氨酰胺代谢;缺氧评分与IL-1信号呈正相关。这说明,细菌在体内和体外促进角质形成细胞缺氧和谷氨酰胺代谢,进而诱导小鼠和人类再生。
最后,研究人员对WIHN下毛囊发育过程中的缺氧和谷氨酰胺代谢进行了研究。为进行
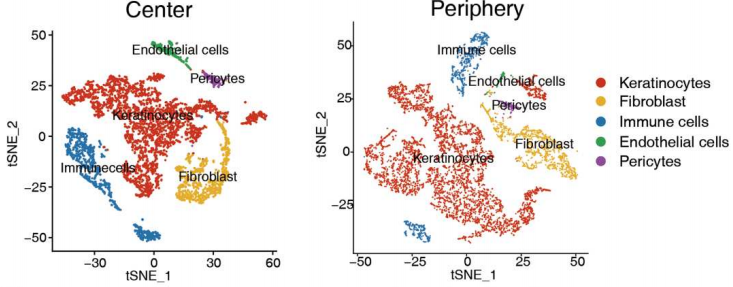
单细胞分辨率下毛囊再生过程中角质形成细胞能量代谢的变化,研究人员区分5种细胞类型:角质形成细胞、成纤维细胞、免疫细胞、内皮细胞和外周细胞,进行了分析。结果显示,IL-1信号在伤口周边缺失,仅在伤口中心角质形成细胞表达。特异性表达模式显示,角质形成细胞释放IL-1,IL-1以自分泌方式发挥作用。
随后研究人员重点研究了角质形成细胞之间的相互作用。结果显示,高WIHN伤口中心的角质形成细胞似乎具有较高的缺氧状态和谷氨酰胺代谢;缺氧基因表达是中央与外周角质形成细胞的主要区别。(图6)这些结果在转录和蛋白水平上验证了高WIHN区的高谷氨酰胺代谢、缺氧和IL-1信号。
图6. WIHN中伤口中心与外周的缺氧和谷氨酰胺代谢升高。
进一步研究的结果显示,高谷氨酰胺代谢的角质形成细胞具有更强的腺苷5′-三磷酸合成、mRNA和蛋白质合成途径,这些途径是毛囊发育所必需的,而谷氨酰胺代谢低的角质形成细胞具有更强的细胞外基质形成、细胞间粘附和细胞连接,这些都是瘢痕组织形成所必需的,很可能抑制WIHN。相比于其他细胞类型,毛囊角质形成细胞的缺氧、谷氨酰胺和谷氨酸代谢及IL-1信号明显更显著。
因为WIHN是一个动态的过程,为了明确表皮分化背景下基底细胞的不同状态,研究人员利用伪时间分析计算了角质形成细胞的分化状态。结果显示,随着毛囊再生细胞的发育,缺氧和谷氨酸的代谢呈总体上升趋势。这说明,缺氧和谷氨酰胺代谢增加可维持角质形成细胞的活性,促进毛囊发育。
该研究是首个全面探究细菌介导毛囊再生的机制研究,创新性地将细菌负荷、缺氧、HIF-1α信号通路、谷氨酰胺代谢、IL-1信号等元素结合,全面阐述了,共生微生物如何诱导创面微环境缺氧,激活HIF-1α信号通路,促进角质形成细胞谷氨酰胺代谢,进而激活下游IL-1β信号通路,促进WIHN。这一研究,对于皮肤毛囊再生的治疗具有重要意义。谷氨酸或IL-1β的添加,可能促进患者伤口愈合,而局部抗生素的使用,则有可能削弱常驻菌群的再生作用。这一研究,将可能为数百万遭受急性和慢性伤口的患者带来改进的、更精确的治疗方法。
参考文献
1. H. Abo, B. Chassaing, A. Harusato, M. Quiros, J. C. Brazil, V. L. Ngo, E. Viennois, D. Merlin, A. T. Gewirtz, A. Nusrat, T. L. Denning, Erythroid differentiation regulator-1 induced by microbiota in early life drives intestinal stem cell proliferation and regeneration. Nat. Commun. 11, 513 (2020).
2. Wang, E. Sweren, H. Liu, E. Wier, M. P. Alphonse, R. Chen, N. Islam, A. Li, Y. Xue, J. Chen, Y. Chen, S. Lee, Y. Wang, S. Wang, N. K. Archer, W. Andrews, M. A. Kane, E. Dare, S. K. Reddy, Z. Hu, E. A. Grice, L. S. Miller, L. A. Garza, Bacteria induce skin regeneration via IL-1β signaling. Cell Host Microbe 29, 777–791.e6 (2021).
3. M. Wickersham, S. Wachtel, T. W. F. Lung, G. Soong, R. Jacquet, A. Richardson, D. Parker, A. Prince, Metabolic stress drives keratinocyte defenses against Staphylococcus aureus infection. Cell Rep. 18, 2742–2751 (2017).