空间表位条形码揭示克隆性肿瘤斑块行为
点评理由:肿瘤内异质性是人类肿瘤的一个重要特征,有助于肿瘤进展和治疗反应。目前的技术在很大程度上不适合精确跟踪肿瘤内的表型和克隆进化。本研究使用组合标记(EpicTags)开发了用于成像的表位并将其与多重离子束成像结合(Epic MIBI),EpicMIBI有助于研究与瘤内异质性相关的细胞内和细胞外过程。
肿瘤癌细胞室中的异质性可能是表观遗传和代谢多样性的结果。多重单细胞成像技术通过同时测量多至数百种蛋白质或RNA,增加了肿瘤内细胞亚型的深度空间表征[1]。特别是多重离子束成像(MIBI)可以捕获翻译后修饰,并以低分辨率测量多种蛋白质[2]。因此,MIBI适用于评估亚细胞区域内的活性和非活性蛋白,同时揭示表型、表观遗传和细胞代谢状态。作为补充,单细胞谱系追踪有助于描绘癌症进化过程中发生的突变[3]。
对显微镜的适应,如用于体细胞突变荧光条形码的CRAINBOW系统,可以提供体内致癌克隆扩增的快照,其应用表明肿瘤异质性不是随机的,而是具有一些可遗传的预定特征。已有研究将蛋白质条形码(Pro Codes)用于通过基于抗体的大规模细胞术跟踪癌细胞[4]。然而,由于空间分辨率或参数有限,细胞跟踪方法受到阻碍。理想情况下,多重单细胞成像将与谱系追踪相结合,以允许对肿瘤微环境的行为和组织进行单个癌细胞的克隆和微扰分析。
近日,由斯坦福大学的Garry P. Nolan和Julien Sage领衔的研究团队在《Cancer Cell》期刊发表重要研究成果,他们使用组合标记(EpicTags)开发了用于成像的表位,并将其与多重离子束成像结合(EpicMIBI),用于组织微环境内的条形码原位跟踪。使用EpicMIBI,他们解剖了小细胞肺癌异种移植模型中细胞谱系和表型的空间成分,观察到混合克隆的突现特性,导致这些模型中神经内分泌和非神经内分泌癌细胞状态的克隆贴片优先扩展。EpicMIBI有助于原位询问与肿瘤内异质性相关的细胞内和细胞外过程。
为了使用表位条形码来区分细胞群,他们首先设计了EpicTag载体(图1A)。接下来,他们使用MIBI来区分细胞颗粒切片中的条码细胞(图1B),产生了由野生型NCI-H82细胞和EpicTag条形码NCI-H82细胞系的混合体组成的细胞颗粒。MIBI鉴定了具有和不具有GFP表达的细胞,并且如预期的那样,在GFP+细胞中特异性地检测到表位的表达(图1C)。每个表位的信号分析显示了具有所有20个条形码的多色图像(图1D、1E)。这些体外数据表明,抗原表位标记蛋白的抗体染色结合多重成像技术,能够保留条形码细胞的空间信息。
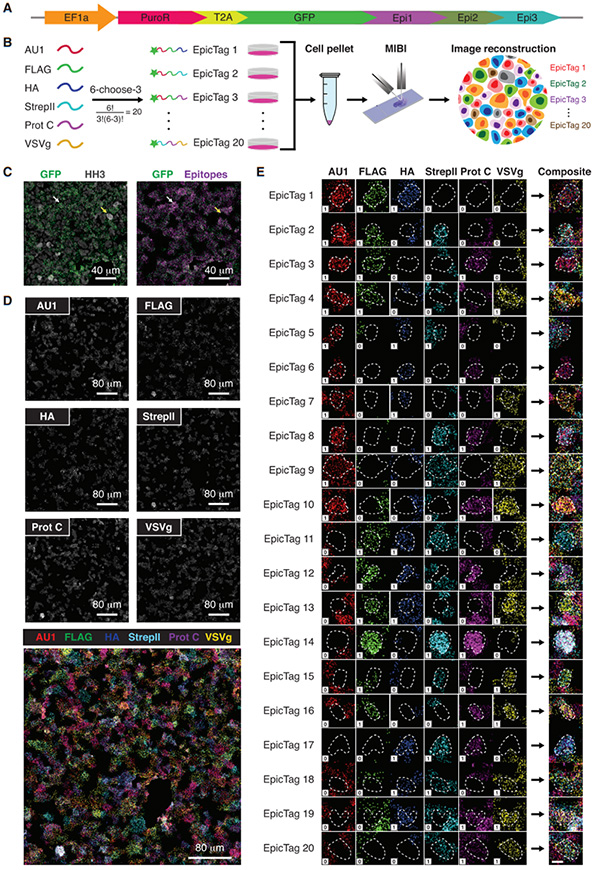
图1 利用基于表位的条形码进行基于图像的特定细胞群识别
接下来,他们通过汇集20个条形码细胞系在小鼠体内产生肿瘤,确定了EpicMIBI策略在体内的潜力。在分离的肿瘤切片上通过MIBI检测表位组合(图2A、2B),表明EpicTag在体内的表达是持久的,EpicMIBI可以作为增殖性小鼠肿瘤中常见祖细胞的代用品。在蛋白质方面,可以用表位组合标记不同的蛋白质,通过混合mCherry+和GFP+癌细胞在小鼠中产生肿瘤,并通过MIBI鉴定了它们(图2C)。他们进一步生成了表达E2和Tag100表位的载体,生长肿瘤并分析切片。进而在体内检测到两个表位,并检测到一起表达多达六个表位的细胞以及GFP或mCherry(图2D和2E)。总之,这些实验证明了EpicTag系统的模块性和可扩展性。
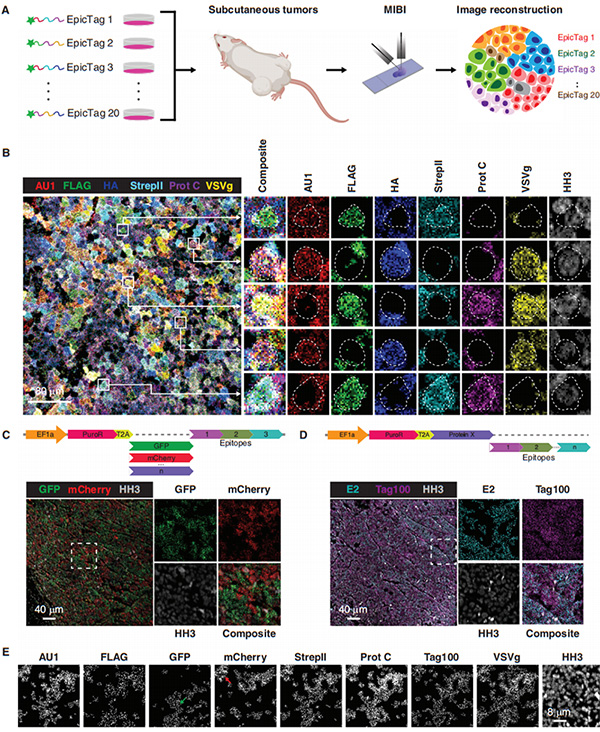
图2 SCLC异种移植物中基于表位的条形码检测
鉴于SCLC-N肿瘤是异质性的,具有NE和非NE特征的癌细胞。研究者使用EpicMIBI研究了属于SCLC-N的NEUROD1high亚型的NCI-H82细胞异种移植物中肿瘤异质性空间组织[5]。他们用抗体组对条形码NCI-H82肿瘤进行染色,以识别肿瘤微环境中的细胞条形码和各种细胞的功能状态(图3A)。所有测试的抗体都产生了预期细胞定位的可检测信号(图3B)。通过GFP表达鉴定NCI-H82细胞,并使用人类特异性线粒体标记进一步确认(图3C)。对于鉴定的每个NCI-H82细胞,分析条形码表达以及NE和非NE分化标记、表观遗传状态和代谢状态(图3D)。他们还鉴定了增殖细胞(Ki-67和组蛋白H3的有丝分裂标志物磷酸-Ser28,pS28 HH3)和DNA损伤细胞(磷酸-Ser139H2AX,γH2AX)(图3E)。总体而言,他们鉴定了八个表型簇(图3F),来自每个表型簇的细胞聚集在一起(图3G),支持聚类结果。
接下来,他们计算了每个表型簇的细胞频率(图3H),并将簇的定位映射到每个tiles瓦片上(图3I)。小鼠基质细胞和内皮细胞(簇1和簇4)存在于所有分析的瓦片中(图3H)。正如对NE细胞系的预期,在所有组合的瓦片中,超75%的细胞是NE细胞(簇6和簇7),这些细胞也是每个单独瓦片中的主要群体(图3H)。H3K27ac高表达的NE细胞(簇7)是所有瓦片中的主要群体,而H3K27ac低表达的NE细胞(簇6)仅在瓦片的子集中以高频存在(图3H和3I)。类似地,非NE细胞(簇2)仅在一些瓦片中以高频出现(图3H和3I)。这些结果表明,在NCI-H82异种移植物中观察到的不同癌细胞状态并非均匀分布,即使在同一肿瘤内也是如此。
从还原论的观点来看,如果肿瘤中存在两种表型A和B,它们可以位于富含A、B或同时含有这两种表型的区域(图3J)。既往研究表明,组织可以表示为细胞邻域(CN)的集合。CN分析框架定义了组织内具有相似周围环境的细胞区域,并能够定量研究细胞位置如何影响功能。研究者在NCI-H82异种移植物中鉴定的表型基于其30个最近的邻居进行聚类,以获得映射到分割图的7个不同的CN。某些CN在具有同型相互作用的细胞中富集:H3K27ac高表达的NE细胞(CN C)、H3K27ac低水平的NE(H3K27aclow)(CN D)和非NE细胞中(CN B)(图3K和3L)。其他CN由几种细胞类型和状态组成。CN F在来自簇3的双阳性细胞中富集,表明该CN中的细胞围绕基质。CN E由H3K27ac高表达和低表达的NE细胞和非NE细胞的混合而成。总体而言,NCI-H82异种移植物中52.9%的细胞位于具有同种型相互作用的CN中。这些分析表明,即使在一个成熟的癌细胞系中,体内也可能出现复杂的空间重排。
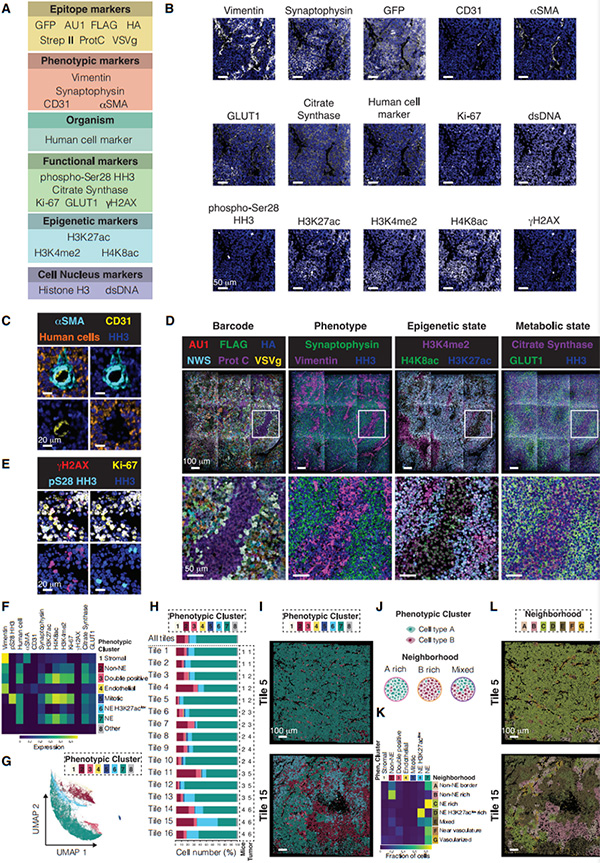
图3 NCI-H82 SCLC异种移植物的空间组织
他们推断,在NCI-H82异种移植物中识别每个癌细胞周围细胞的祖先将为塑造肿瘤结构的过程提供见解。通过整合基于细胞和像素的去条形码方法,他们开发了克隆肿瘤图,以表示条形码细胞的空间位置(图4A和STAR方法)。他们识别了NCI-H82异种移植物中20种可能的表位组合(图4B、4C),基于细胞和像素的去编码方法识别了所有条形码,每个去编码的细胞系都表达了预期的表位(图4D),这表明基于表位图像的计算解构识别了肿瘤内癌细胞群的共同祖先。
然后,他们对NCI-H82异种移植物数据集进行去编码,以获得每个瓦片的克隆肿瘤图。在单细胞提取和合并后获得了每个条形码的百分比(图4E)。克隆肿瘤图传达了瘤内细胞-细胞相互作用的局部复杂性图像,在这种克隆肿瘤图谱中可以推断出某些影响肿瘤生长的局部癌细胞行为。共享条形码的癌细胞预计将被检测为单独分布的细胞,并被分为不同大小的克隆肿瘤块(图4F)。此外,预期具有相同条形码的单个细胞和克隆肿瘤贴片彼此靠近并散布在肿瘤中(图4F)。这种分布预计与具有不同条形码的其他癌细胞的分布一致(图4F)。这些分析表明,癌细胞在NCI-H82异种移植物中不是随机分布的,并且倾向于形成克隆斑块。
肿瘤内细胞的空间分布可以看作是成对距离的网格(图4F)。他们发现某些斑块在同一条形码的其他斑块附近共存(图4G)。对于数据集中的每个瓦片,他们应用滑动窗口方法来量化克隆斑块富集的区域,肿瘤的某些区域富含单个细胞和较小尺寸的斑块(图4H)。这些结果表明,NCI-H82异种移植物的克隆生长在空间上并不局限于单个斑块,而是取决于局部环境。
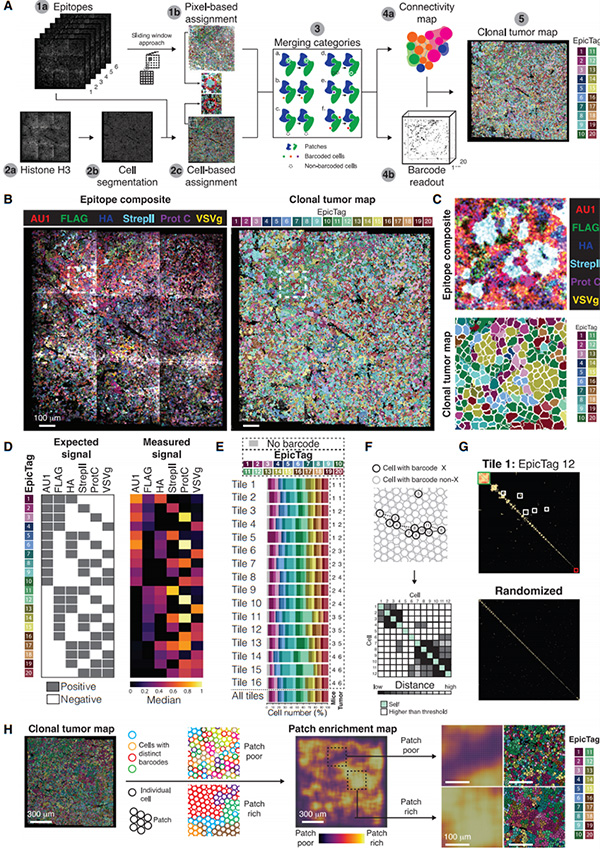
图4 一种去编码策略识别克隆性肿瘤斑块
可以为条形码NCI-H82异种移植物数据集中的每个细胞提取三层基本信息:细胞所属的等基因细胞系(EpicTag条形码)、其类型和状态(表型簇)以及表达的功能标记(图5A)。空间成分提供了关于每个细胞周围细胞(即CN)的类型和状态、周围癌细胞(即肿瘤斑块)的克隆性推断以及标记物的亚细胞位置的深层信息(图5A)。为了进行综合数据可视化,他们绘制了所有CN中非NE、NE H3K27aclow和NE细胞状态(分别为簇2、簇6和簇7)的所有条形码细胞的8个标记物的表达。每个癌细胞状态和CN中条形码之间的标记物表达的变异系数表明,在其各自的同型CN中的三种主要癌细胞状态(非NE、NE和NE H3K27aclow)以及在其同型CNs中的簇外标记物表达相似(图5B)。这些分析显示了跨簇和CN的条形码之间良好的标记表达一致性。
EpicTag条形码的NCI-H82细胞产生各种大小的斑块。他们询问细胞总数是否与每个条形码和表型簇的斑块数量和大小增加相关。对于NE细胞,细胞和斑块的数量在条形码细胞系中强烈相关(图5C)。相反,非NE细胞的相关性较低(图5C)。这表明NE和非NE斑块的扩增机制不同。
非NE细胞(簇2)产生的斑块少于NE细胞的斑块(簇7)(图5C)。线性模型的斜率比率表明,对于NE细胞(簇7),斑块中的细胞比非NE细胞多1.8倍(簇2),这表明非NE细胞包含在较大的斑块中(图5D)。事实上,与NE和其他细胞状态相比,非NE的斑块明显更大(图5E)。这些数据表明,具有非NE表型的NCI-H82细胞在细胞分裂后保持在一起,并倾向于形成更大的斑块。在所有条形码群体中均鉴定出非NE细胞(簇2)(图5E),但与NE对应物(簇7)相比,六个条形码细胞系产生了更大的非NE(簇2,图5E)。这些结果表明,体内生长的NCI-H82细胞本质上主要是NE细胞,但大面积的非NE细胞有时会“稳定”并在未知刺激下扩张。
他们推测,这些大块非NE细胞的产生可能归因于增殖增加。,正如预期,对非静止细胞标记物Ki-67的分析表明,有丝分裂细胞(簇5)具有最高水平的Ki-67表达(图5F)。在非NE细胞(簇2)中,Ki-67水平显著增加,静止细胞的数量显著减少(图5F和5G)。相反,Ki-67表达、静止细胞数量和NE细胞斑块大小之间没有显著相关性(簇7)(图5F和5G)。NE H3K27aclow细胞(簇6)具有低增殖指数(图5F和5G),突出了与NE细胞的功能差异。值得注意的是,在较大的非NE(簇2)斑块中,柠檬酸合成酶的表达显著升高,但在非NE细胞(簇2和簇7)中,不同斑块大小的GLUT1水平相似(图5H和5I),表明与较大的非NE斑块中的糖酵解相比,柠檬酸循环的活性升高。最后,他们注意到,非NE和NE细胞的较大斑块显示出较少的DNA损伤(图5J)。这些结果表明一旦形成小斑块,DNA损伤减少、代谢变化和增殖增加都能导致较大的非NE克隆斑块扩大。
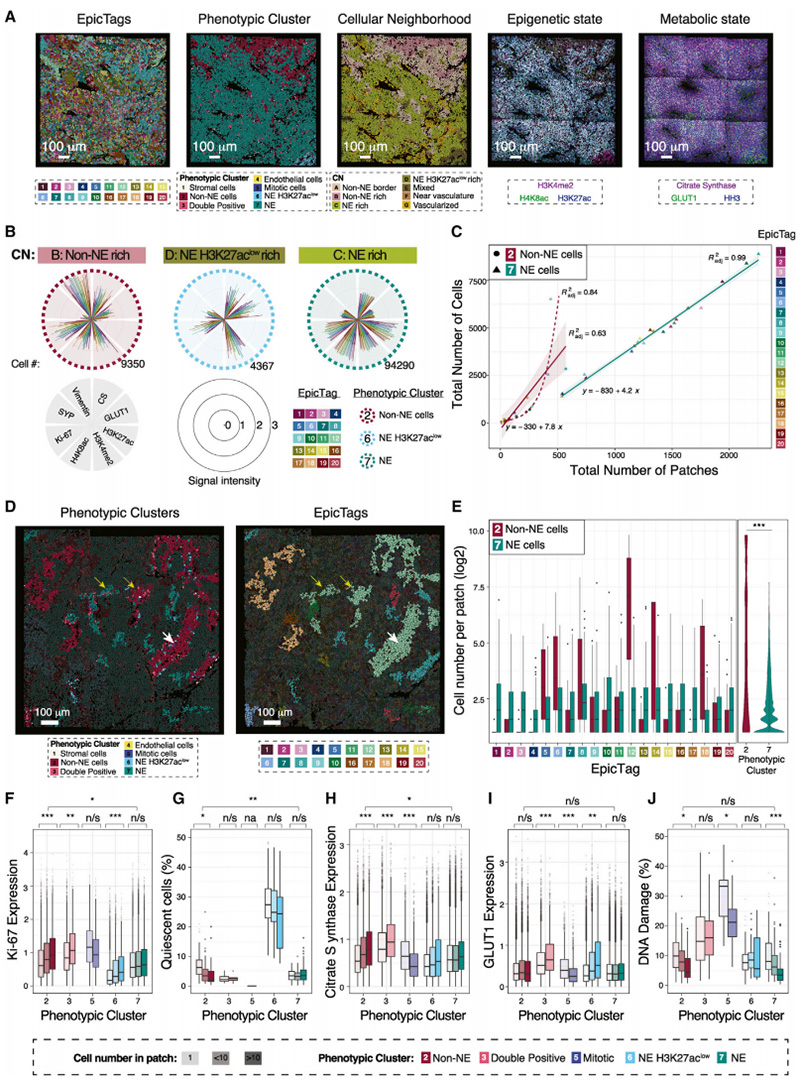
图5 非神经内分泌SCLC细胞形成大斑块,增殖指数增加,DNA损伤减少
总之,该研究使用条形码系统和亚细胞多重蛋白成像来识别小细胞肺癌模型中的克隆行为。原位跟踪条形码癌细胞的能力,加上其空间环境的特征,为理解肿瘤演变开辟了新的途径。利用在亚细胞水平上追踪具有不同遗传修饰组的复杂群体的能力,未来将能够确定基因型和表型与细胞邻域以及细胞间和多克隆行为的因果关系。
参考文献
[1] Chen A, Liao S, Cheng M, Ma K, Wu L, Lai Y, Qiu X, Yang J, Xu J, Hao S, Wang X, Lu H, Chen X, Liu X, Huang X, Li Z, Hong Y, Jiang Y, Peng J, Liu S, Shen M, Liu C, Li Q, Yuan Y, Wei X, Zheng H, Feng W, Wang Z, Liu Y, Wang Z, Yang Y, Xiang H, Han L, Qin B, Guo P, Lai G, Muñoz-Cánoves P, Maxwell PH, Thiery JP, Wu QF, Zhao F, Chen B, Li M, Dai X, Wang S, Kuang H, Hui J, Wang L, Fei JF, Wang O, Wei X, Lu H, Wang B, Liu S, Gu Y, Ni M, Zhang W, Mu F, Yin Y, Yang H, Lisby M, Cornall RJ, Mulder J, Uhlén M, Esteban MA, Li Y, Liu L, Xu X, Wang J. Spatiotemporal transcriptomic atlas of mouse organogenesis using DNA nanoball-patterned arrays. Cell. 2022 May 12;185(10):1777-1792.e21. doi: 10.1016/j.cell.2022.04.003. Epub 2022 May 4. PMID: 35512705.
[2] Keren L, Bosse M, Thompson S, Risom T, Vijayaragavan K, McCaffrey E, Marquez D, Angoshtari R, Greenwald NF, Fienberg H, Wang J, Kambham N, Kirkwood D, Nolan G, Montine TJ, Galli SJ, West R, Bendall SC, Angelo M. MIBI-TOF: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 2019 Oct 9;5(10):eaax5851. doi: 10.1126/sciadv.aax5851. PMID: 31633026; PMCID: PMC6785247.
[3] Quinn JJ, Jones MG, Okimoto RA, Nanjo S, Chan MM, Yosef N, Bivona TG, Weissman JS. Single-cell lineages reveal the rates, routes, and drivers of metastasis in cancer xenografts. Science. 2021 Feb 26;371(6532):eabc1944. doi: 10.1126/science.abc1944. Epub 2021 Jan 21. PMID: 33479121; PMCID: PMC7983364.
[4] Wroblewska A, Dhainaut M, Ben-Zvi B, Rose SA, Park ES, Amir ED, Bektesevic A, Baccarini A, Merad M, Rahman AH, Brown BD. Protein Barcodes Enable High-Dimensional Single-Cell CRISPR Screens. Cell. 2018 Nov 1;175(4):1141-1155.e16. doi: 10.1016/j.cell.2018.09.022. Epub 2018 Oct 18. PMID: 30343902; PMCID: PMC6319269.
[5] Rudin CM, Poirier JT, Byers LA, Dive C, Dowlati A, George J, Heymach JV, Johnson JE, Lehman JM, MacPherson D, Massion PP, Minna JD, Oliver TG, Quaranta V, Sage J, Thomas RK, Vakoc CR, Gazdar AF. Author Correction: Molecular subtypes of small cell lung cancer: a synthesis of human and mouse model data. Nat Rev Cancer. 2019 Jul;19(7):415. doi: 10.1038/s41568-019-0164-2. Erratum for: Nat Rev Cancer. 2019 May;19(5):289-297. PMID: 31175338.